SEQUENCE PLANTS AND ANIMALS WITH CONFIDENCE
과학자들이 광범위한 식물 및 동물을 연구하는 것에 있어
생물학적 질문에 답하는 데 SMRT sequencing이
어떻게 도움이 되는지 알아보십시오.


크고 복잡한 genome은 polyploidy 및 높은 수준의 heterozygosity와 같은 독특한 문제를 제시합니다. PacBio는 이러한 문제를 극복하는 데 필요한 높은 정확도와 충분히 긴 길이의 read를 제공합니다.

CALLING ALL VARIANT TYPES
PacBio의 매우 정확하고 길이가 충분히 긴 HiFi read는 genome의 모든 변이를 포괄적으로 감지합니다. 향상된 변이 검출 능력으로 유전자와 원인 변이를 새롭게 발견할 수 있고 유전과 관심있는 표현형을 연결하는 능력을 향상시킵니다.
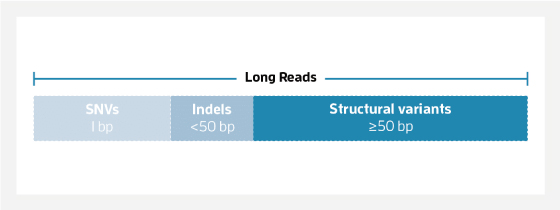
50bp 이하의 SNV (Single nucleotide variants), Indel (insertions and deletions) 50bp 이상의 SV (structural variants) 등 모드 종류의 변이를 PacBio의 매우 정확하고 충분히 길이가 긴 HiFi read로 분석할 수 있습니다.
|
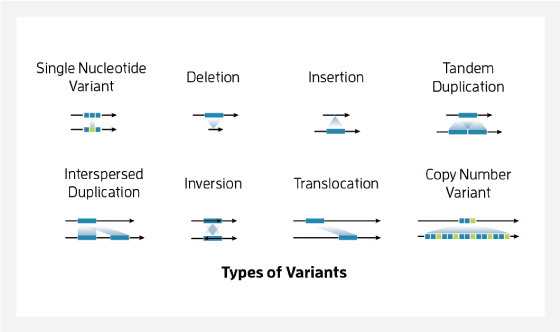
Sequel IIe 시스템에서 생성된 HiFi read를 사용하면 연간 100 ~ 1000 개의 genome 변이를 감지할 수 있습니다. HiFi read는 매핑하기 어려운 반복 영역을 포함하여 SNV (single nucleotide variants), indels, SV (structural variants), CNV( copy number variants) 분석에 높은 정밀도와 재현율을 제공합니다. |
Tracing plant immunity with resistance genes

resistance gene을 통해 식물 면역을 밝히고자 하는 연구에 대해 소개합니다. 식물 면역 연구는 수용체 – microbe-associated molecular patterns (MAMP)을 인식하는 표면 수용체 및 pathogen effector (e.g. nucleotide-binding leucine-rich repeat receptors, NLRs)를 감지하는 세포 내 수용체 – 에 초점을 맞추고 있습니다.
Arabidopsis thaliana 에서 nucleotide-binding leucine-rich repeat receptors(NLR)의 거의 완전한 종 전체 데이터베이스 생성결과에 대해 발표하였습니다. 그들이 얻은 시퀀스를 통해 핵심 NLR 요소를 정의하고 연관된 도메인의 다양성을 차트로 작성, 새로운 도메인 아키텍처를 설명하고 non-core NLR에서 다형성의 유무를 평가하고 특성화되지 않은 NLR을 A. thaliana Col-0 reference genome에 매핑할 수 있었습니다.
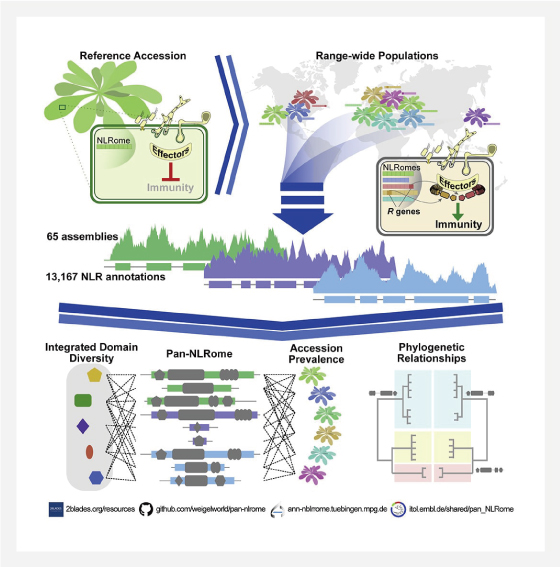
Van de Weyer, A.-L., et al. (2019). Cell
A Species-Wide Inventory of NLR Genes and Alleles in Arabidopsis thaliana.
DOI : 10.1016/j.cell.2019.07.038
수백 개의 NLR 유전자가 flowering plant에서 발견됩니다. 이 NLR 유전자는 병원체 인식 후 일어나는 cell death를 컨트롤하는 resistosomes라는 물질을 형성한다고 여겨지고 있어 새로운 병원체 저항성을 만들기 위한 후보물질로 이 resistosomes를 연구하고 있습니다. 그러나 매우 polymorphic한 특성을 가지고 있는 gene family, allelic patterns 그리고 구조적 variation과 광범위한 copy-number variation을 가진 gene cluster 등 극복해야 할 여러 부분들이 있습니다.
두 연구팀이 resistance gene enrichment sequencing (RenSeq)과 SMRT sequencing을 결합하여 이러한 문제점들을 성공적으로 극복했습니다. 두 팀 중 한 팀은 토마토 식물의 NLRome을 연구하였습니다. RenSeq는 선택적으로 토마토의 NLRs를 찾아 시퀀싱 할 수 있었지만, NLRs의 highly repetitive한 sequences와 physical clusters가 있는 부분들은 완전히 해결할 수 없어 SMRT sequencing으로 전환을 하였고 SMRT RenSeq의 다양한 장점을 확인하였습니다. 예를 들면, SMRT RenSeq는 whole genome sequencing의 능률적인 대안이면서, 비용적으로도 효율적입니다. 또한, NLRs의 complexity와 NLR clusters도 해결할 수 있었습니다. NLR의 high-quality gene model은 crop engineering 연구를 위한 자원으로 활용될 수 있습니다.
Structural variation and adaptation in cattle

Wai Yee Low et al. (2019) biorxiv
DOI: doi.org/10.1101/720797
연구 성과 & haplotype-resolved genome assembly 이 연구는 high-quality haplotype-resolved genome assembly를 통해 알아본 taurine cattle(타우린 소)와 indicine cattle 간의 생산성 관련 형질 차이 및 환경 적응력 형질 차이에 대해 설명합니다. 이것은 급변하는 환경에서 소의 생산성을 보장해줄 중요한 phenotypic traits를 발견하는데 도움을 줍니다. Taurine cattle과 indicine cattle의 haplotype-resolved genome assemblies는 SNP부터 SV (structural variant)에 이르는 genetic variants를 찾는데 사용되었고, 이는 아종의 특성을 구별할 수 있게 하였습니다. 예를 들어, indicine cattle의 경우, fatty acid desaturase(지방산불포화효소)의 extra copy가 positively selected 되어 indicine cattle이 열과 가뭄에 적응할 수 있게 한다고 생각하고 있습니다. Haplotype-resolved genomes의 또 다른 이점은 이들이 diploid transcriptome profile에서 allele-specific expression을 더 쉽게 찾고 해석하는 데 사용될 수 있다는 것입니다. 여기서, allele-specific 및 novel transcripts는 haplotype-resolved genomes에 mapping된 PacBio Iso-Seq reads를 사용하여 찾을 수 있었습니다.
그 외 사용된 methods/technologies - 본 연구에서는 PacBio reads, Hi-C reads, RNA-Seq, Iso-Seq, Illumina paired-end reads 등의 기술이 사용되었습니다. - trio binning metho를 사용하여 contig를 생성하였습니다. - Hi-C reads, Illumina paired-end reads를 통해 haplotype-resolved scaffolding을 제공하고 assembly error를 발견할 수 있었습니다. - Long PacBio reads는 Sanger sequencing으로 생성된 이전의 cattle assembly에 비해 repeat resolution이 향상시켰습니다. - Full-length transcripts의 identification 및 phasing은 PacBio Bioconda의 Isoseq 3.1.0 소프트웨어를 사용하여 처리된 Iso-Seq 데이터를 통해 수행하였습니다. - Scaffolds의 gap들은 haplotype-specific PacBio subreads를 사용하여 메울 수 있었습니다.
|
Revealing genetic mysteries of marine life
|
해양 생물의 비밀을 밝혀내기 위해 필요한 SMRT sequencing |
많은 과학자들은 다양한 해양 생물의 genome과 transcriptome의 매우 정확한 long-read sequencing을 위해 PacBio SMRT sequencing을 사용합니다. 이것은 해양 생물들이 어떻게 적응하고 진화하는지 알려주고, 보존 노력에 기여하며, 보다 효율적인 aquaculture practices (수산양식업)을 구축하는 등 해양 생물학의 모든 영역에 대한 정보를 제공합니다.
적응 및 진화 SMRT sequencing을 사용하여 진화와 관련된 molecular mechanism에 대해 연구하는 것은 적응과 관련한 유전학에 새로운 통찰력을 가지게 합니다. . 예를 들어 C. aceratus와 O. minor의 genome과 transcriptome을 sequencing한 것은 그들이 온도나 산소량과 같은 environmental stressor에 어떠한 메커니즘으로 적응하는지 알게 하였습니다.
보존 해수 온도 상승이 산호 생태계에 미치는 영향 – 예를 들어 산호 표백 (coral bleaching)과 전염병 확산 등 – 을 이해하기 위해 다양한 SMRT sequencing 방법들을 사용되고 있습니다. 산호 genome, transcriptome 및 관련 microbiome에 대한 연구는 산호의 stress response에 대한 새로운 통찰력을 제공합니다.
양식(aquaculture) 효율적인 양식업은 많은 해양 생물들의 생존 뿐만 아니라 증가하는 인구의 수요를 충족하는 데에도 매우 중요합니다. PacBio sequencing을 사용하여 면역 기능, 질병 저항력 그리고 성 결정(sex determination)과 관련한 genetics를 이해하려 하고 있습니다. 예를 들어, Pacific white shrimp 게놈에서 면역에 영향을 미치는 부분의 isoform-level reference transcriptome을 생성하기 위해 RNA sequencing 기술이 행해지며, 이는 이 생물의 특이한 면역 체계에 대해 이해할 수 있게 합니다. |
Zhou Y., et. al. (2019) Nature Plants
DOI: 10.1038/s41477-019-0507-8
구조 변이(SV)는 식물 genome의 아직 많이 밝혀져 있지 않은 영역입니다. SV의 유형과 크기, 개체 간의 분포, 특히 population dynamics에 대해서는 알려진 바가 거의 없습니다. 이러한 역학을 이해하는 것은 표현형에 대한 SV의 기여와 genome 전체의 연관성에서 인과적 유전 변이로서의 가능성을 구별하는 것 모두에 중요합니다.
구조 변이를 식별하고 클론으로 증식된 포도나무 품종과 야생 조상 종을 교배하는 데 있어 진화 유전체학을 연구한 논문이 발표되었습니다.
구조 변이를 분류하기 위해 7개 유전자 중 1개가 SV를 기반으로 하는 hemizygous 인 heterozygous Chardonnay genome을 assembly하였습니다. Whole genome, long-read 및 short read alignment로 Chardonnay와 Cabernet Sauvignon geome 간의 통합 비교를 사용하여 SV 검출을 모집단 샘플로 확장했습니다. 강력한 purifying selection이 SV에 대해 작용하지만 특히 역전 및 전위 이벤트에 대해 작용한다는 것을 발견하였습니다. 그럼에도 불구하고 SV는 클론으로 증식된 계통에서 열성 이형 접합체로 축적되고 또한 야생 및 재배 포도 사이의 genome divergence의 outlier region을 정의하여 재배종에서의 역할을 제시합니다. Outlier region 영역에는 독립적인 크고 복잡한 inversion이 있는 성별 결정 영역과 berry 색상 영역이 포함되어 있으며, 집중적인 표현형 진화를 주도했습니다.
Exploring crustacean genomes for viruses

Satoshi Kawato et al. (2019)
DOI: 10.1128/JVI .01144-18
흰반점증후군 바이러스 (White Spot Syndrome Virus; WSSV)는 새우 산업에서 전 세계적으로 심각한 갑각류 감염 병원체입니다. 이 논문은 숙주의 genome 데이터에 숨겨진 새로운 WSSV 연관종 (nimavirus라고도 불림) genome을 사용하여 WSSV의 진화 역사를 추적하는 것을 목표로 합니다.
본 연구에서는 14개의 갑각류 (숙주) genome을 조사했고, 5개의 새로운 nimaviral genome0을 찾아냈습니다. WSSV와 그 친족의 게놈을 비교한 결과, WSSV는 자신을 치명적인 병원체로 만드는데 주요 원인이 될 수 있는 특유의 숙주-바이러스 상호 작용 관련 유전자를 가지고 있는 것으로 나타났습니다. 게다가, 곤충을 감염시키는 바이러스 단백질 동족체 (homolog)의 보존은 다른 곤충 감염 DNA virus와 갑각류 감염 Nimaviridae의 공통적인 phylogenetic origin을 암시합니다. 이처럼 본 연구는 특성이 잘 알려지지 않았던 이전의 갑각류 바이러스 계열을 재정의하고 심각한 새우 병원체를 예고했었던 오래된 genomic event들을 밝혀낼 수 있습니다.
Smoking out structural variants in the cannabis genome

이 웨비나에서는 Type II (THCA와 CBDA를 생성하는) 식물의 대마초 게놈 (cannabis genome)에 대해 설명합니다. 또한, PacBio의 long-read sequencing이 어떻게 high-quality plant genomics research를 가능하게 하는 지와 full-length transcript sequencing이 cannabis reference genome을 annotation 하는 것, 그리고 다양한 종류의 대마초 식물에서 일어나는 cannabis synthesis(대마초 합성)에 대한 새로운 통찰력 등 cannabis genomics의 최신 정보를 공유한다.
________________________________________