SEQUENCE PLANTS AND ANIMALS WITH CONFIDENCE
과학자들이 광범위한 식물 및 동물을 연구하는 것에 있어
생물학적 질문에 답하는 데 SMRT sequencing이
어떻게 도움이 되는지 알아보십시오.
크고 복잡한 genome은 polyploidy 및 높은 수준의 heterozygosity와 같은 독특한 문제를 제시합니다. PacBio는 이러한 문제를 극복하는 데 필요한 높은 정확도와 충분히 긴 길이의 read를 제공합니다.

Insect Biology
질병 예방 방법을 개발하고, 기본 생물학을 이해하고, 집단 또는 개체를 사용하여 해충 방제를 개선합니다.
Crop Improvements
작물의 건강을 개선하고, 형질 선택을 수행, 번식 효율성을 향상시키는 새로운 생물학을 발견하십시오.
Animal Health and Breeding
동물 유전학을 조사하여 유전, 면역학 및 가축화 패턴을 고해상도로 연구합니다.
Marine Biodiversity
해양 생태계의 깊숙한 곳으로 뛰어 들어 진화를 연구하고 개발 및 기후 변화의 과제를 해결하십시오.
Conservation
지원 번식, 야생 동물 관리 및 지속 가능성을 지원하기 위해 위험에 처한 종의 유전적 다양성을 조사합니다.
PLANT AND ANIMAL WHOLE GENOME SEQUENCING

PacBio HiFi read는 긴 길이 (최대 25kb)와 높은 정확도 (> 99.9 %)를 모두 제공하여 가장 복잡한 genome의 연속적이고 완전하며 정확한 de novo 게놈 어셈블리를 빠르고 경제적으로 생성합니다.

Agricultural pest rapidly sequenced
Sarah B Kingan et. Al. (2019) GigaScience
DOI : 10.1093/gigascience/giz122
PacBio Sequel II를 이용한 High-quality genome assembly
- High-quality genome assembly는 침입 종 (invasive species), 질병 벡터 (disease vector) 또는 멸종위기 종 보존 노력 등을 위한 프로젝트에 필요한 접근법입니다.
- SMRT (Single Molecular, Real-Time) 시퀸싱 기술의 Sequel II System은 훨씬 저렴한 비용으로 보다 높은 정확도와 처리량으로 long-reads를 생성할 수 있으며, 생성된 Data는 DNA 함량 및 시퀀스 복잡도 전반에 걸쳐 최소한의 sequencing bias를 가집니다.
- 단 한 마리의 현장 채집된 해충 침입종 (invasive species) 꽃매미 spotted lanternfly 에서 한 개의 SMRT Cell과 하나의 sequencing library만으로 2.25Gb 게놈의 sequencing 및 high-quality de novo assembly를 진행하였습니다. 또한 lanternfly 외에도 2개의 세균성 체내 공생체 (bacterial endosymbiont)의 완전한 게놈을 곧바로 생성할 수 있었습니다.
Genome of the ice worm, a glacier dwelling annelid
Webinar: Small Bodies, Big Genomes: Overcoming Large DNA Input Requirements for Long-Read Genome Assembly
이 웨비나에서는 소형 유기체(small-bodied organism)의 high-quality genome assembly 생성하기 위한, DNA requirement 부터 library preparation과 sequencing에 이른 low DNA input workflow 에 대해 설명하고 있습니다. 대략 1.6 Gb 크기의 게놈을 가지며 빙하에서 사는 North American ice worm (Mesenchytraeus solifugus)에 이 workflow를 적용하였고, 혹독한 환경에 적응할 수 있는 적응력을 살펴볼 수 있습니다.
Sequence Even the Smallest Organisms with Confidence
연속적이고 정확한 genome은 곤충 생물학을 이해하고 질병 매개체 및 농업 해충을 통제하는 데 필수적인 도구입니다. 그러나 크기가 작은 유기체를 연구하는 데는 몇 가지 주요 과제가 있습니다.
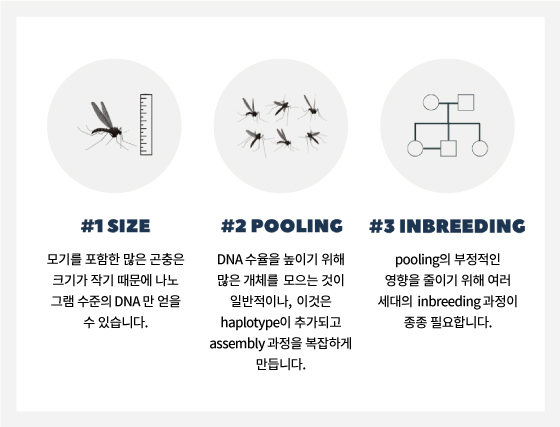
PacBio에는 이러한 문제를 극복하는 데 도움이 되는 두 가지 워크 플로우가 있습니다.
• Low DNA Input Workflow
– a HiFi sequencing approach requiring 300 ng -400 ng of input DNA
• Ultra-Low DNA Input Workflow
– A HiFi sequencing approach, with the addition of an amplification step, requiring 5 ng of input DNA
Minding the Gaps:
International Team Creates First Two Gapless Rice Reference Genomes

|
쌀은 최초로 완성된 농작물 genome이었지만, 완벽하게 완성된 적은 없었습니다. International Team은 마침내 gap과 누락된 sequence를 채워, 전 세계적으로 공급되는 xian/indica 쌀 품종의 gap-free 참조 유전체 두 개를 생성하였습니다.
PacBio의 CLR 및 HiFi sequencin을 포함한 정확한 long-read 시퀀스 데이터 와 높은 커버리지, 그리고 여러가지 어셈블리 방법들을 통해 어셈블리 gap들을 모두 메울 수 있었습니다. Hi-C와 Bionano map을 사용하여 어셈블리의 퀄리티를 입증했으며, FISH와 ChIP-Seq assay를 사용하여 centromere의 위치와 1차 구조(primary structure)를 찾아내었습니다. |
저자는 "높은 정확도와 repeat-spanning reads를 가진 PacBio HiFi long-read sequencing은 복잡한 heterozygous 영역과 centromere의 어셈블리를 위한 훌륭한 자원이었다"고 말하고 있습니다.
새로운 어셈블리는 99.88%의 BUSCO score와 LTR 어셈블리 지수 (LTR assembly index; LAI) 데이터를 저장하였는데 이는 gold / platinum reference genome의 기준을 충족하였습니다. 또한, 기존의 어셈블리에서는 몇 십개의 rRNAs가 발견되었던 반면에, 이번에는 1,500개 이상을 찾을 수 있었습니다. Assembly에서 마지막으로 채워진 gap들은 모두 centromere 영역에 있었는데, 이는 특히 더 큰 genome일수록 재조합(recombination)이 크게 감소하기 때문입니다.
이러한 rice reference genome은 잡종강세(heterosis) 연구와 다른 기본적인 또는 응용된 연구를 위한 핵심적이고 포괄적인 모델을 발전시키는 데 도움이 될 것이며, 식물생물학에서의 reference genome의 새로운 기준이 되는 길로 이끌 것입니다.
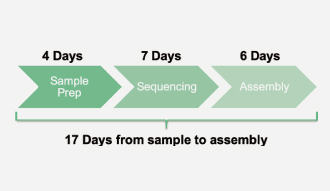
Sequencing the California redwood

PacBio의 new HiFi reads로 하는 sequencing은 매우 정확한 long-read를 생성합니다. 최소 99%의 정확도로 최대 25kb read를 생성 할 수 있습니다. 이러한 long-read의 길이는 짧은 repeats를 포함할 수 있고 high accuracy로 크고 복잡한 repeats를 구별할 수 있기 때문에 balance를 이루며 complex genome의 assembly를 성공적으로 해낼 수 있습니다.
|
PacBio HiFi reads를 사용하여 California redwood (27 Gb giant genome, hexaploidy)를 assembly하는 데 단지 17일이 걸렸습니다. – sample preparation 4일, sequencing 7일, assembly 6일. 하나의 assembly는 1.92Mb의 contig N50을 가진 것으로 예상했던 게놈의 거의 두 배 크기였습니다. |
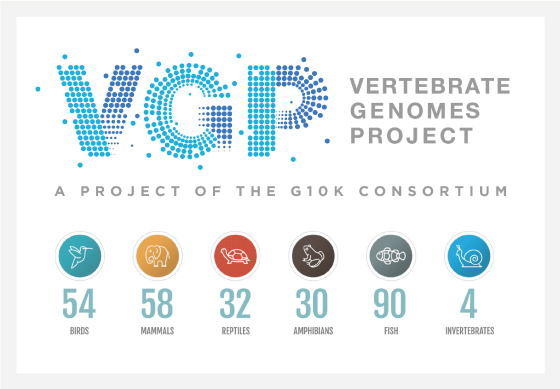
Consortium releases 100 high-quality genomes
보전 유전학자(Conservation Geneticist) 들의 international consortium은 척추동물 게놈 프로젝트 (VGP)의 일환으로 100개의 high-quality assembly들을 공개하였습니다. 100개의 게놈은 멸종 위기에 처한 vaquita porpoise(바키타 돌고래)처럼 보전과 과학적 관심이 필요한 다양한 종들의 유전자를 포함하고 있습니다.
|
지구상에 현존하는 모든 척추동물– 전체 70,000 종-의 chromosomal-level genome assemblies를 만들기 위한 프로젝트의 일환으로, 전세계에서 마지막으로 남아 있는 바키타 돌고래의 DNA가 보존되고 해독되었습니다. 샌디에이고 Zoo Global의 한 Conservation Genetics 팀은 "다른 멸종 위기에 처한 porpoise에 속한 종들에게 도움이 될 수 있는 유용한 정보가 나올 수 있을 것"이라고 언급하였습니다. |
|
보존을 넘어, 몇몇 종의 genome은 진화의 과정을 이해하는 데에도 유용합니다. 예를 들어, 흔히 '살아있는 공룡'으로 불리는 장수거북(Dermochelys coriacea)은 독특한 생리학적 적응 능력을 가진 고대 혈통으로 멸종될 위험이 매우 높습니다. VGP genome은 남아있는 이 종의 유전적 다양성을 연구하는데 사용될 것입니다. 또한, 다른 개체군과의 유전적 혼합을 통해 과잉 교배를 막을 수 있게 하는 전위(translocation)처럼, 장수거북을 보존하는 방법들을 알기 위해서도 VGP genome이 사용될 것입니다. |
닭과 같은 동물의 genome은 흔하고 평범해 보이지만, 농업과 생의학 연구에 필수입니다. "Trio binning method”를 통해, 부모의 DNA를 사용하여 자식 염색체의 DNA 서열을 분리, 하나의 개체로부터 (각각 어머니와 아버지로부터 온) 두 개의 게놈을 assembly합니다. 이러한 trio-based assembly는 parentally-inherited된 DNA를 분리하는데 있어 non-trio based assembly보다 40-60% 더 우수합니다.